コロナウイルス研究
新型コロナウイルスの病原性を明らかにする病理学
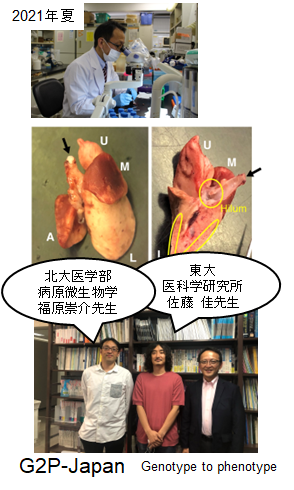
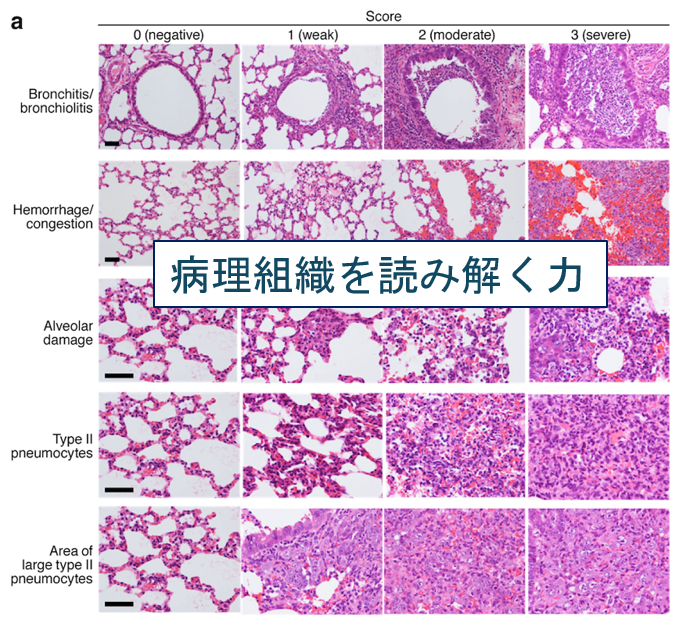
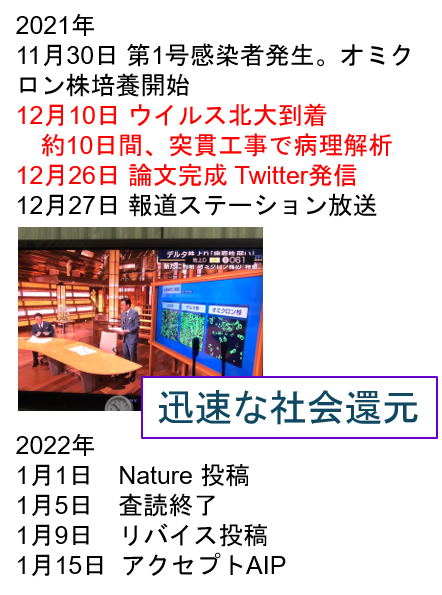
未知のウイルス感染症が出現した時に、病理組織解析が大きな力となりました。2021年コロナ禍の真っ最中、北大の微生物の福原先生から、コロナウイルスを感染せたハムスターの肺炎像をみてくださいという依頼をうけました。G2P-Japan(代表佐藤佳教授)の進める研究とのことでした。ハムスターの解析の経験はありませんでしたが、まずは切り出して標本を作成して(①)、お盆を一週間返上して朝から晩までHE標本を見直して組織評価のスコアリング法を確立しました(②)。デルタ株の研究論文が受理されて、この病理組織解析が役に立ちそうだということで、オミクロンが出現して病原性が社会問題問題となった際には、12月10日に北大にウイルスが到着して、寝る間もなく2週間突貫工事で解析をして結果を出しました。チーフの佐藤先生が12月26日Twitter配信して翌日報道ステーションで報道となりました。自分自身はウイルス学者でもなく呼吸器病理専門でもありませんが、よりどころになったのは日々のCPCで呼吸器内科の先生とのdiscussionでした。未知の病原体が出現した際に、組織病理学の力が病態解明に極めて重要である、ということがあらためてわかりました。若手病理医・研究者の皆様にお伝えできればと思います。
- ハムスターモデル
- SARS-CoV-2コロナウイルス肺炎の炎症scoring
- オミクロン株の病原性の解明と発信

腫瘍病理学教室とG2P-Japanの共同研究論文
Shinya Tanaka: Co-corresponding author
- Saito A, Irie T, Suzuki R, Maemura T, Nasser H, Uriu K, Kosugi Y, Shirakawa K, Sadamasu K, Kimura I, Ito J, Wu J, Iwatsuki-Horimoto K, Ito M, Yamayoshi S, Loeber S, Tsuda M, Wang L, Ozono S, Butlertanaka E P, Tanaka Y L, Shimizu R, Shimizu K, Yoshimatsu K, Kawabata R, Sakaguchi T, Tokunaga K, Yoshida I, Asakura H, Nagashima M, Kazuma Y, Nomura R, Horisawa Y, Yoshimura K, Takaori-Kondo A, Imai M, Chiba M, Furihata H, Hasebe H, Kitazato K, Kubo H, Misawa N, Morizako N, Noda K, Oide A, Suganami M, Takahashi M, Tsushima K, Yokoyama M, Yuan Y, Tanaka S, Nakagawa S, Ikeda T, Fukuhara T, Kawaoka Y, Sato K, and The Genotype to Phenotype Japan C. Enhanced fusogenicity and pathogenicity of SARS-CoV-2 Delta P681R mutation. Nature, 602, 300-306, 2022.
- Suzuki R, Yamasoba D, Kimura I, Wang L, Kishimoto M, Ito J, Morioka Y, Nao N, Nasser H, Uriu K, Kosugi Y, Tsuda M, Orba Y, Sasaki M, Shimizu R, Kawabata R, Yoshimatsu K, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Sawa H, Ikeda T, Irie T, Matsuno K, Tanaka S, Fukuhara T, and Sato K. Attenuated fusogenicity and pathogenicity of SARS-CoV-2 Omicron variant. Nature, 603, 700-705, 2022.
- Kimura I, Yamasoba D, Tamura T, Nao N, Suzuki T, Oda Y, Mitoma S, Ito J, Nasser H, Zahradnik J, Uriu K, Fujita S, Kosugi Y, Wang L, Tsuda M, Kishimoto M, Ito H, Suzuki R, Shimizu R, Begum M M, Yoshimatsu K, Kimura K T, Sasaki J, Sasaki-Tabata K, Yamamoto Y, Nagamoto T, Kanamune J, Kobiyama K, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Shirakawa K, Takaori-Kondo A, Kuramochi J, Schreiber G, Ishii K J, Hashiguchi T, Ikeda T, Saito A, Fukuhara T, Tanaka S, Matsuno K, and Sato K. Virological characteristics of the SARS-CoV-2 Omicron BA.2 subvariants, including BA.4 and BA.5. Cell, 185, 3992-4007.e3916, 2022.
- Yamasoba D, Kimura I, Nasser H, Morioka Y, Nao N, Ito J, Uriu K, Tsuda M, Zahradnik J, Shirakawa K, Suzuki R, Kishimoto M, Kosugi Y, Kobiyama K, Hara T, Toyoda M, Tanaka Y L, Butlertanaka E P, Shimizu R, Ito H, Wang L, Oda Y, Orba Y, Sasaki M, Nagata K, Yoshimatsu K, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Kuramochi J, Seki M, Fujiki R, Kaneda A, Shimada T, Nakada T A, Sakao S, Suzuki T, Ueno T, Takaori-Kondo A, Ishii K J, Schreiber G, Sawa H, Saito A, Irie T, Tanaka S, Matsuno K, Fukuhara T, Ikeda T, and Sato K. Virological characteristics of the SARS-CoV-2 Omicron BA.2 spike. Cell, 185, 2103-2115.e2119, 2022.
- Ito J, Suzuki R, Uriu K, Itakura Y, Zahradnik J, Kimura K T, Deguchi S, Wang L, Lytras S, Tamura T, Kida I, Nasser H, Shofa M, Begum M M, Tsuda M, Oda Y, Suzuki T, Sasaki J, Sasaki-Tabata K, Fujita S, Yoshimatsu K, Ito H, Nao N, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Yamamoto Y, Nagamoto T, Kuramochi J, Schreiber G, Saito A, Matsuno K, Takayama K, Hashiguchi T, Tanaka S, Fukuhara T, Ikeda T, and Sato K. Convergent evolution of SARS-CoV-2 Omicron subvariants leading to the emergence of BQ.1.1 variant. Nat Commun, 14, 2671, 2023.
- Tamura T, Ito J, Uriu K, Zahradnik J, Kida I, Anraku Y, Nasser H, Shofa M, Oda Y, Lytras S, Nao N, Itakura Y, Deguchi S, Suzuki R, Wang L, Begum M M, Kita S, Yajima H, Sasaki J, Sasaki-Tabata K, Shimizu R, Tsuda M, Kosugi Y, Fujita S, Pan L, Sauter D, Yoshimatsu K, Suzuki S, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Yamamoto Y, Nagamoto T, Schreiber G, Maenaka K, Hashiguchi T, Ikeda T, Fukuhara T, Saito A, Tanaka S, Matsuno K, Takayama K, and Sato K. Virological characteristics of the SARS-CoV-2 XBB variant derived from recombination of two Omicron subvariants. Nat Commun, 14, 2800, 2023.
- Tamura T, Yamasoba D, Oda Y, Ito J, Kamasaki T, Nao N, Hashimoto R, Fujioka Y, Suzuki R, Wang L, Ito H, Kashima Y, Kimura I, Kishimoto M, Tsuda M, Sawa H, Yoshimatsu K, Yamamoto Y, Nagamoto T, Kanamune J, Suzuki Y, Ohba Y, Yokota I, Matsuno K, Takayama K, Tanaka S, Sato K, and Fukuhara T. Comparative pathogenicity of SARS-CoV-2 Omicron subvariants including BA.1, BA.2, and BA.5. Commun Biol, 6, 772, 2023.
- Fujita S, Plianchaisuk A, Deguchi S, Ito H, Nao N, Wang L, Nasser H, Tamura T, Kimura I, Kashima Y, Suzuki R, Suzuki S, Kida I, Tsuda M, Oda Y, Hashimoto R, Watanabe Y, Uriu K, Yamasoba D, Guo Z, Hinay A A, Jr., Kosugi Y, Chen L, Pan L, Kaku Y, Chu H, Donati F, Temmam S, Eloit M, Yamamoto Y, Nagamoto T, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Suzuki Y, Ito J, Ikeda T, Tanaka S, Matsuno K, Fukuhara T, Takayama K, and Sato K. Virological characteristics of a SARS-CoV-2-related bat coronavirus, BANAL-20-236. EBioMedicine, 104, 105181, 2024.
- Ito H, Tamura T, Wang L, Mori K, Tsuda M, Suzuki R, Suzuki S, Yoshimatsu K, Tanaka S, and Fukuhara T. Involvement of SARS-CoV-2 accessory proteins in immunopathogenesis. Microbiol Immunol, 68, 237-247, 2024.
- Tamura T, Ito H, Torii S, Wang L, Suzuki R, Tsujino S, Kamiyama A, Oda Y, Tsuda M, Morioka Y, Suzuki S, Shirakawa K, Sato K, Yoshimatsu K, Matsuura Y, Iwano S, Tanaka S, and Fukuhara T. Akaluc bioluminescence offers superior sensitivity to track in vivo dynamics of SARS-CoV-2 infection. iScience, 27, 109647, 2024.
- Tamura T, Mizuma K, Nasser H, Deguchi S, Padilla-Blanco M, Oda Y, Uriu K, Tolentino J E M, Tsujino S, Suzuki R, Kojima I, Nao N, Shimizu R, Wang L, Tsuda M, Jonathan M, Kosugi Y, Guo Z, Hinay A A, Jr., Putri O, Kim Y, Tanaka Y L, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Saito A, Ito J, Irie T, Tanaka S, Zahradnik J, Ikeda T, Takayama K, Matsuno K, Fukuhara T, and Sato K. Virological characteristics of the SARS-CoV-2 BA.2.86 variant. Cell Host Microbe, 2024.
- Tsujino S, Deguchi S, Nomai T, Padilla-Blanco M, Plianchaisuk A, Wang L, Begum M M, Uriu K, Mizuma K, Nao N, Kojima I, Tsubo T, Li J, Matsumura Y, Nagao M, Oda Y, Tsuda M, Anraku Y, Kita S, Yajima H, Sasaki-Tabata K, Guo Z, Hinay A A, Jr., Yoshimatsu K, Yamamoto Y, Nagamoto T, Asakura H, Nagashima M, Sadamasu K, Yoshimura K, Nasser H, Jonathan M, Putri O, Kim Y, Chen L, Suzuki R, Tamura T, Maenaka K, Irie T, Matsuno K, Tanaka S, Ito J, Ikeda T, Takayama K, Zahradnik J, Hashiguchi T, Fukuhara T, and Sato K. Virological characteristics of the SARS-CoV-2 Omicron EG.5.1 variant. Microbiol Immunol, 2024.
